DOI:https://doi.org/10.46502/issn.2710-995X/2021.6.03
Análisis de desempeño de segmentación sobre imágenes de muestras de sangre periférica
Performance analysis of segmentation on images of peripheral blood samples
Resumen
La sicklemia es una enfermedad con alta incidencia en la población cubana. Se caracteriza por la deformación del eritrocito y puede estudiarse empleando técnicas automatizadas para análisis de imágenes de sangre, que dependen de la calidad de los bordes detectados. En este trabajo se propone un estudio comparativo sobre el desempeño de cuatro métodos de segmentación (Umbralización, Mean-Shift, Level- Set y Chan-Vese) aplicados a este tipo de imágenes. La experimentación permitió demostrar la superioridad del método Mean-Shift, los resultados fueron evaluados empleando métricas de media, varianza, distancia de Baddeley e índice de Jaccard y Dice.
Palabras clave: Mean-Shift, métricas, muestras de sangre periférica, segmentación y sicklemia.
Abstract
Sickle Cell Disease have a high incidence in the Cuban population. It is characterized by the deformation of the erythrocyte and can be studied using automated techniques for analysis of blood images, which depend on the quality of the edges detected by segmenting the cells. In this work we propose a comparative study on the performance of four segmentation methods (Threshold, Mean-Shift, Level-Set and Chan-Vese) applied to this type of images. The experimentation allowed to demonstrate the superiority of the Mean- Shift method, the results were evaluated using metrics of mean, variance, Baddeley distance and Jaccard and Dice index.
Key Words: Mean-Shift, metrics, peripheral blood samples, segmentation, sickle cell disease.
Introducción
Los eritrocitos son células de la sangre encargadas del transporte del oxígeno a los órganos y tejidos, y del dióxido de carbono para su eliminación, estos pueden presentar un gran número de variaciones en su tamaño, forma y color. Específicamente, la variación en su forma constituye uno de los primeros indicadores de la existencia de determinadas enfermedades en los seres humanos. Una de estas enfermedades es la anemia drepanocítica, sicklemia o enfermedad de las células falciformes, enfermedad genética que provoca la deformación de los glóbulos rojos, los cuales adoptan forma de hoz o media luna a la vez que se tornan rígidos pues pierden flexibilidad debido a la falta de oxígeno que presentan. Esta enfermedad se caracteriza por un incremento de la susceptibilidad a las infecciones, retraso del crecimiento y desarrollo de crisis vaso oclusivas dolorosas lo que provoca la aparición de nuevas lesiones inflamatorias en los pacientes que la padecen (Fernández, Pérez, Fragoso & Rivero, 2012). En la actualidad es la hemoglobinopatía más frecuente, por esta razón la Organización Mundial de la Salud (OMS) considera la misma como un problema de salud a nivel mundial, principalmente en países con ascendencia africana (WHO, 2001), entre los cuales se encuentra Cuba. Estudios estadísticos han demostrado que la frecuencia de portadores en nuestro país oscila entre el 3 y el 8 % en las diferentes regiones (Fernández et al, 2012), destacándose la provincia de Guantánamo como la de mayor incidencia, donde la tasa de portadores se eleva hasta cerca de 51 por cada 1000 habitantes, constituyendo todavía un problema social y de salud pública.
En tal sentido, en los laboratorios de hematología del Hospital General Docente “Dr. Agostinho Neto” de la provincia de Guantánamo, para el seguimiento a pacientes con esta patología se valoran exámenes complementarios entre los que están el hemograma y los frotis de sangre periférica. Estas muestras de sangre son observadas en el microscopio por parte del especialista en hematología, este proceso resulta demasiado complejo y tedioso pues consume mucho tiempo ya que el análisis de las muestras es agotador y propenso a la ocurrencia de errores, cuya frecuencia crece a medida que aumenta el número de muestras a analizar. Además, la eficiencia del reconocimiento se ve afectada por otros factores subjetivos como la experiencia del especialista en hematología, que es el factor determinante en la emisión de un criterio valorativo sobre la deformación celular que presenta el paciente, pues la dificultad del proceso complejiza obtener un criterio cuantitativo de eritrocitos deformados en la sangre, únicamente se aporta una valoración personal del experto en hematología sobre el nivel de daño del paciente en este sentido.
En aras de obtener un criterio cuantitativo sobre el nivel de deformación celular presente en las muestras de sangre de pacientes con anemia drepanocítica, el procesamiento de imágenes es un paso fundamental para esta tarea, y en este caso la detección eficiente de los bordes y regiones de los cuerpos que forman parte de las muestras de sangre mediante el proceso de segmentación constituye una etapa crítica, debido a que el posterior procesamiento que pueda realizarse para el análisis de las formas a partir de estos bordes segmentados depende de la calidad con que se obtienen los mismos. En el caso específico del análisis automatizado de muestras de sangre, en el período 2014 – 2020 fueron empleados diferentes algoritmos de segmentación, entre los más populares estuvieron: Umbralización, Mean-Shift, Level-Set y Chan-Vese. También se pudo constatar que la selección de los métodos para la realización de la segmentación de imágenes de muestras de sangre carece de una adecuada evaluación comparativa mediante el uso de métricas de segmentación que garanticen una mejor elección de los algoritmos, para obtener óptimos resultados en la selección de las regiones de interés de estas imágenes. Para valorar la calidad de la segmentación obtenida fueron utilizadas cinco métricas de evaluación de similitud: la media y la varianza (métricas de evaluación estadísticas), distancia de Baddeley (métrica de evaluación geométrica) y el índice de Jaccard y Dice (estadísticos que miden el grado de similitud entre dos conjuntos).
Este proceso además está condicionado a las características de la imagen, tamaño, niveles de ruido, iluminación y textura, estas dependen en gran medida del proceso de preparación de las muestras y adquisición de las imágenes; las imágenes a procesar son muestras de sangre completa que presentan gran cantidad de artefactos lo que incrementa la dificultad al realizar la segmentación. Hasta el momento, no se ha realizado un estudio valorativo de la efectividad de este tipo de métodos de segmentación en imágenes de muestras de sangre obtenidas bajo condiciones de preparación en laboratorios de entidades de salud pública de nuestro país, que, por nuestras propias características, asociadas muchas veces a las propiedades de los recursos disponibles para su preparación, en muchos casos, tienen una alta presencia de objetos extraños, zonas con iluminación diferente, etc.
De esta forma se pretende realizar el análisis de imágenes médicas de muestras de sangre de pacientes con anemia drepanocítica obtenidas bajo las condiciones de los laboratorios clínicos del Hospital General Docente “Dr. Agostinho Neto” de la provincia de Guantánamo y se pretende determinar de los métodos de segmentación Umbralización, Mean-Shift, Level-Set y Chan-Vese cuál brinda una mejor respuesta ante este tipo de imágenes médicas. Por tanto, este trabajo investigativo está dirigido a la evaluación de desempeño de los métodos de segmentación antes mencionados, mediante el empleo de métricas adecuadas de valoración para la segmentación.
Marco Teórico
Para el desarrollo de la investigación se considera el período comprendido entre el año 2016 y el 2020. Los términos de búsqueda definidos para esta investigación son: eritrocitos, muestras de sangre, enfermedades en la sangre, anemia drepanocítica y sicklemia. Luego de realizar una búsqueda breve, se encontraron algunos artículos de los que se extrajeron nuevas palabras clave. Estas palabras clave son: forma de eritrocitos, morfología, segmentación de imágenes de muestras de sangre. Combinando las palabras claves encontradas con los primeros términos de búsqueda, formamos la cadena de consulta final de la siguiente manera: análisis de imágenes sobre muestras de sangre con anemia drepanocitica, preocesamiento de imagenes y segmentación de imágenes de muestras de sangre con drepanocitosis. Para esta revisión, se escogieron artículos y referencias publicadas a partir de 2016, considerando los que tienen los siguientes criterios: artículos con objetivos de investigación bien definidos, con detalles sobre el proceso de segmentación, evaluación de la segmentación, la extracción y presentación de los datos en análisis de muestras de sangre de pacientes con anemia drepanocitica y que se hayan publicado en revistas o conferencias escritas en español o inglés.
Los siguientes tipos de documentos fueron excluidos: artículos bajo el proceso de discusión o que fueron remitidos a otros tipos de componentes de la sangre y no incluye los eritrocitos. Encuentros informales de literatura (sin preguntas de investigación definidas, sin proceso de búsqueda, sin proceso definido de extracción o análisis de datos). No se valoraron artículos que, aunque se refieren a patologías relacionadas con eritrocitos, no afectan como tal la forma de estos (por ejemplo: la malaria).
Metodología
Para enfrentar el proceso de determinar qué método de segmentación de imágenes posee mejor prestación al ser aplicados en imágenes de muestras de sangre de pacientes con anemia drepanocítica, la metodología aplicada se dividió en cuatro pasos fundamentales:

Figura 1. Proceso de determinación del mejor método de segmentación. Fuente: Elaboración propia.
1.Obtención de contornos y regiones de prueba: Esta tarea se realizará de forma manual por parte del especialista en hematología de conjunto con el equipo de trabajo que desarrolla la propuesta, la misma tiene el objetivo elegir adecuadamente cada uno de los cuerpos de interés real, dentro de las imágenes de muestras de sangre de pacientes con anemia drepanocitica. Las imágenes empleadas para el estudio, fueron adquiridas en los laboratorios de hematología del Hospital General Docente “Dr. Agostinho Neto” de la provincia de Guantánamo, las mismas pertenecen a muestras de sangre de varios pacientes con anemia drepanocítica en estado de crisis. Se tomaron en total de 60 imágenes de una resolución 1024x768 px a partir de las muestras obtenidas en un microscopio Leika con objetivo de aumento 100x, empleando una cámara Kodak de 8.0 megapíxeles con cañón AF 3X OPTICAL no profesional. A continuación, se describen los pasos lógicos a realizar:
I. Seleccionar la imagen de muestra de sangre.
II. Seleccionar los objetos de interés dentro de la imagen, desechando cuerpos extraños que no representen eritrocitos.
III. Generación de contornos de los objetos seleccionados dentro de la imagen.
IV. Generación de regiones de los objetos seleccionados dentro de la imagen.
En la Figura 2 se muestra un ejemplo de la obtención del contorno y la región de prueba con la participación de un especialista.

Figura 2. Obtención de contornos y regiones de prueba. Fuente: Elaboración propia.
a) Imagen de muestra de sangre.
b) Selección de los objetos de interés.
c) Contorno de prueba.
d) Región de prueba.
1. Obtención de contornos y regiones mediante métodos de segmentación: Esta tarea tiene el objetivo ejecutar los métodos de segmentación propuestos, sobre las imágenes de muestras de sangre de pacientes con anemia drepanocitica. A continuación, se describen los pasos lógicos a realizar:
I. Seleccionar la imagen de muestra de sangre.
II. Convertir la imagen seleccionada a escala de grises.
III. Seleccionar el método de segmentación a utilizar.
IV. Generación automática de contornos de los objetos en la imagen seleccionada mediante el método de segmentación escogido.
V. Generación automática de regiones de los objetos en la imagen seleccionada mediante el método de segmentación escogido.
A continuación, se muestra una imagen seleccionada, su equivalente a escala de grises y la obtención de los contornos y regiones generados mediante los métodos de segmentación escogidos.

Figura 3. Obtención de contornos y regiones mediante métodos de segmentación. Fuente: Elaboración propia.
a) Imagen de muestra de sangre. b) Imagen de muestra de sangre a escala de grises. c) Umbralización. d) Mean-Shift. e) Level-Set. f) Chan-Vese.
La segmentación es el proceso mediante el cual se particiona una imagen en regiones que son homogéneas con respecto a una o más características bajo cierto criterio, esto se logra identificando los píxeles que pertenecen a una misma clase (González & Woods, 2018). Un gran número de técnicas se han propuesto para el proceso de segmentación, las que a continuación serán descritas representan el mayor porcentaje de uso en investigaciones científicas llevadas hasta la fecha.
Descripción de los métodos de segmentación:
Umbralización: El método de umbralización hace una clasificación de la imagen en dos clases de pixeles. Este se considera apropiado precisamente para aplicar en imágenes como las de muestras de sangre, en las que los valores de intensidad de los objetos estén fuera del rango de los valores de intensidad del fondo. Básicamente podemos explicar la umbralización como sigue:
Tenemos una imagen 𝒇(𝒙, 𝒚) compuesta por objetos luminosos y fondo oscuro, de tal forma que los pixeles de los objetos y los del fondo se pueden separar por su intensidad en dos grupos. Una forma obvia de separar los objetos del fondo es definir un umbral t que separe estos dos grupos. Entonces cualquier punto (𝒙, 𝒚) para el cual 𝒇(𝒙, 𝒚) ≥ 𝒕 es llamado un punto objeto, en caso contrario es llamado un punto fondo (Rahmat, Wulandari, Faza, Muchtar & Siregar, 2018). En otras palabras, la imagen 𝒈(𝒙, 𝒚) se define como:

La determinación del umbral adecuado para una imagen dada es un factor crítico de la segmentación. Un método que permite calcular el valor de umbral automáticamente es el método de Otsu (Vasundhara & Preetham, 2017), que es el empleado en este trabajo.
Mean-Shift: El método de las medias desplazadas (Mean-Shift, en inglés) es una aproximación de agrupamiento donde cada objeto se mueve al área más densa en su proximidad (Jinghua, Jie, Juan & Lihui, 2011). El mismo se basa en un esquema iterativo que considera que el espacio de datos es una función de densidad de probabilidad muestreada y para cada punto del conjunto de datos, se encuentra la moda más cercana, definiendo una región alrededor de ese punto y encontrando su media, cambiando la situación de la media actual a la nueva (Shift), repitiéndose el proceso hasta que converja. En cada iteración del algoritmo se realizan sobre cada uno de los puntos de la imagen los siguientes pasos:
1. Se define una ventana de tamaño arbitrario. 2. Se ubica un punto 𝑥 de la imagen en el centro de la ventana y se calcula la media correspondiente al conjunto de puntos pertenecientes a la misma. Para el cálculo de la media se utiliza la ecuación:

donde: • 𝑁(𝑥) es el conjunto de puntos que se encuentran dentro de la ventana. 𝑥 es el punto seleccionado. • ℎ es el ancho de ventana que representa el rango de influencia que se le da a cada elemento. • 𝑘 es una función núcleo utilizada para aproximar una función de densidad de probabilidad subyacente del conjunto de datos. 3.Se reemplaza el valor del punto por el valor obtenido en el cálculo de la media.
Level-Set: El método de segmentación Level-Set es un método numérico para controlar la evolución de contornos y superficies. En lugar de manipular el contorno directamente, el contorno se introduce como la curva de nivel cero dentro de una función de orden superior llamada función Level-Set 𝜓(𝑋, 𝑡) (Xin, Renjie & Shengdong, 2012). Esta función se hace evolucionar bajo el control de una ecuación diferencial. En cualquier momento puede obtenerse el contorno envolvente extrayendo la curva de nivel cero 𝜓((𝑋), 𝑡) = {𝜓(𝑋, 𝑡) = 0} a partir de la salida obtenida. La siguiente ecuación representa la ecuación diferencial genérica para una función de Level-Set; cada algoritmo de este tipo particularizará de una manera distinta dicha ecuación diferencial.

Donde 𝐴 es el término de advenimiento, 𝑃 el termino de propagación (expansión) y 𝑍 un modificador espacial para la curvatura media 𝑘. Los escalares 𝛼, 𝛽, y 𝛾 son pesos relativos a la influencia de cada término en la evolución. En un caso de uso típico, un contorno es inicializado por el usuario y es luego evolucionado hasta que se ajusta a la forma anatómica de la estructura de la imagen. Chan-Vese: El algoritmo Mumford-Shah es usado para establecer un criterio óptimo para la segmentación de imágenes en subregiones. Una imagen es modelada como una función de suavidad por partes. La función penaliza la distancia entre el modelo y la imagen original, la falta de suavidad en el modelo dentro de las subregiones y el tamaño de los límites de las subregiones. Mediante la minimización de esta función se obtiene el mejor resultado de la segmentación. Sea 𝑓 que denota una imagen dada en escala de grises en el dominio 𝛺 a ser segmentado, se aproxima la imagen 𝑓 por una función de suavidad por partes 𝜇 como la solución de un problema de minimización, donde 𝐶 es el borde de una curva dada.
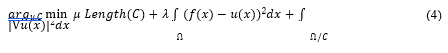
Comparado con el modelo Mumford-Shah, la principal diferencia de Chan-Vese es que se adiciona un nuevo término que penaliza el área encerrada y una mayor simplificación de 𝜇, permitiendo que 𝜇 solo tome dos valores (Getreuer, 2012), o sea que el resultado de la segmentación será una imagen binaria:

Donde 𝐶 es el borde de una curva dada y 𝑐1 y 𝑐2 son los valores de 𝜇 adentro y afuera de 𝐶 respectivamente. El método Chan-Vese consiste en encontrar entre todas las 𝜇 la que mejor se aproxima a 𝑓:

El primer término asegura la regularidad de 𝐶 mediante la penalización del tamaño de los límites, el segundo término penaliza el área dentro de 𝐶 controlando su tamaño y el tercero y el cuarto penalizan las discrepancias entre 𝑓 y 𝜇.
1. Aplicación de las métricas a los contornos y regiones obtenidos: Esta fase tiene como objetivo ejecutar las métricas, sobre los contornos y regiones de prueba con respecto a los contornos y regiones generados por los métodos de segmentación en la imagen seleccionada. A continuación, se describen los pasos lógicos a realizar:
I. Seleccionar los contornos y regiones de prueba, en la imagen de muestra de sangre seleccionada.
II. Seleccionar los contornos y regiones generados por los métodos de segmentación, en la imagen de muestra de sangre escogida.
III. Seleccionar la métrica de evaluación.
IV. Aplicar la métrica sobre los contornos y regiones de prueba con respecto a los contornos y regiones generada por los métodos de segmentación, en la imagen de muestra de sangre.
V. Mostrar los resultados de la métrica.
Los métodos de evaluación de segmentación de imágenes pueden ser clasificados en métodos analíticos y métodos empíricos. Los métodos analíticos tratan los algoritmos de segmentación directamente y están dirigidos a examinar su principio de funcionamiento; mientras que, los métodos empíricos juzgan la imagen segmentada de acuerdo con un criterio predefinido (métodos no supervisados), o comparando con una imagen de referencia (métodos supervisados), para evaluar indirectamente el rendimiento de los algoritmos. La evaluación empírica es prácticamente más eficaz y la más utilizada, los métodos empíricos se clasifican en: grupo de métodos de bondad (Goodness methods, en inglés), y grupo de métodos de diferencias (Discrepancy methods, en inglés). Estos grupos de métodos usan diferentes criterios empíricos para juzgar el rendimiento de los algoritmos de segmentación. Los Métodos de Bondad pueden realizar la evaluación sin la ayuda de imágenes de referencia, mientras que los Métodos de Diferencias necesitan alguna imagen de referencia para arbitrar la calidad de la segmentación (Gil, 2011).
En este trabajo se emplean métodos empíricos de diferencias los cuales comparan la imagen segmentada o la imagen de salida contra una imagen de referencia (mejor resultado esperado de la segmentación) y usan sus diferencias para evaluar el rendimiento de los algoritmos de segmentación. Estos métodos tratan de determinar cuán lejos está la imagen segmentada de la imagen de referencia. Un valor más alto de la medida de diferencias implicaría un error más grande en la imagen segmentada relativa a la imagen de referencia, y esto indica que el algoritmo de segmentación aplicado tiene un rendimiento más bajo.
Descripción de las métricas de evaluación:
Medidas de media y varianza: Para evaluar un método de detección de contornos se utilizan dos criterios estadísticos característicos de la detección de error: la media y la varianza (Panduro, 2010).

Donde 𝑑(𝑖) es la distancia entre el 𝑖𝑡ℎ píxel de la segmentación 𝐼𝑠 y el pixel más cercano en el contorno de prueba 𝐼𝑡. La primera medida da una idea de la distancia media que existe entre los pixeles del contorno de prueba y los de la segmentación 𝐼𝑠, aunque puede darse el caso en el que una segmentación 𝐼𝑠 tenga gran cantidad de pixeles muy cercanos a 𝐼𝑠 y otro grupo de pixeles muy lejanos, o sea que exista dispersión en los pixeles y aun así obtener una buena media. Sin embargo, esto no pasa con la varianza, la cual penaliza los pixeles más alejados haciendo que la medida de similitud crezca mucho si hay pixeles muy dispersos. Como consecuencia de esto se tiene que si una segmentación aporta un buen valor para la media y uno malo para la varianza entonces los pixeles de esta están dispersos y no es una buena segmentación. Otro problema que tienen estas dos medidas es que no penaliza formas abruptas en la segmentación. En la Figura 4 se muestra el resultado de la segmentación 𝐼𝑠 y el contorno de prueba 𝐼𝑡.

Figura 4. Forma abrupta en los contornos. Fuente: Elaboración propia.
Para obtener la media y la varianza en el punto 𝑃 o cualquier punto en la vecindad de este, el pixel más cercano en 𝐼𝑡 será el punto 𝐾 y no quedará reflejado en el resultado la forma abrupta que tiene el contorno 𝐼𝑡, o sea, en el resultado de la medida no existirá diferencia, aunque el contorno muestre esta característica. Para enfrentar esta situación es usada la distancia de Baddeley.
Distancia de Baddeley: Sea 𝑋 un dominio común de dos resultados de segmentación 𝐼1 y 𝐼2

Donde 𝐼1 y 𝐼2, corresponden al conjunto de pixeles del contorno de la segmentación. 𝑑(𝑥, 1) es la distancia del píxel 𝑥 al píxel más cercano en el contorno de la imagen 𝐼 y 𝑃 > 1
Índice de Jaccard: El índice de Jaccard mide el grado de similitud entre dos conjuntos, sea cual sea el tipo de elementos. La formulación es la siguiente:

El mismo siempre toma valores entre 0 y 1, correspondiente este último a la igualdad total entre ambos conjuntos (Altinsoy et al, 2019).
Índice de Dice: Este coeficiente es un estadístico utilizado para comparar la similitud de dos muestras, pero también puede ser visto como una medida de similitud sobre conjuntos que varía de 0 a 1 en dependencia de la similitud (Altinsoy et al, 2019):

Los métodos de segmentación propuestos, así como las métricas de evaluación de segmentación fueron implementadas en el software MatLab R2017a 64 bit para Windows, fue empleada una GUIDE (editor de interfaces de usuario - GUI) lo que permite interactuar y llevar a cabo parte del proceso requerido. Todas las pruebas fueron realizadas en una computadora con microprocesador Intel(R) Core (TM) I7- 4800MQ a 2.7 GHz con 8 Gb de memoria RAM.
4. Evaluación de resultados: En la sección de Resultados y discusión, se realiza un mejor análisis de esta última fase del proceso para de determinar el mejor método de segmentación.
Resultados y discusión
A continuación, se muestran en formato tabular y gráfico, los valores obtenidos por cada una de las métricas de evaluación de métodos de segmentación. Es válido retomar el significado de cada una de estas medidas al ser aplicadas:
1. Media: El valor obtenido por este criterio estadístico representa cuán lejanos se encuentran los píxeles de los contornos obtenidos por los algoritmos, respecto a los pixeles de los contornos de prueba, por lo que, a menores valores, mejor es la segmentación obtenida. 2. Varianza: La medida obtenida representa la dispersión a la que se encuentran los píxeles de los contornos obtenidos por los algoritmos, respecto a los pixeles de los contornos de prueba, pero en este caso penalizada la dispersión existente entre los pixeles con valores muy elevados, denotando una peor segmentación para los métodos en los que este valor sea más alto. 3. Distancia de Baddeley: Esta es una medida de distancia más realista entre los pixeles de los contornos obtenidos por los algoritmos, respecto a los pixeles de los contornos de prueba aportados por los especialistas. Esta métrica penaliza formas abruptas entre los contornos y a menor valor de esta distancia mejor es el desempeño del método de segmentación. 4. Índice de Jaccard y Dice: Los valores generados por estas dos métricas, permiten obtener una medida de similitud entre los pixeles pertenecientes a las regiones generadas por los algoritmos de segmentación y los pixeles de las regiones que forman parte de las imágenes aportados por los especialistas. Dichas medidas se expresan en valores entre 0-1, denotando los valores cercanos a cero la disparidad entre los conjuntos y valores cercanos a uno la igualdad entre estos.
Tabla 1.
Valores de la media y varianza para cada método de segmentación.

Fuente: Elaboración propia.

Figura 5. Representación gráfica de los valores de la media para cada método de segmentación. Fuente: Elaboración propia.

Figura 6. Representación gráfica de los valores de varianza para cada método de segmentación. Fuente: Elaboración propia.
Tabla 2. Valores de la distancia de Baddeley para cada método de segmentación

Fuente: Elaboración propia.

Figura 7. Representación gráfica de los valores de distancia de Baddeley para cada método de segmentación. Fuente: Elaboración propia.
Tabla 3. Valores del índice de Jaccard para cada método de segmentación.

Fuente: Elaboración propia.

Figura 8. Representación gráfica de los valores del índice de Jaccard para cada método de segmentación. Fuente: Elaboración propia.

Figura 9. Representación gráfica de los valores del índice de Dice para cada método de segmentación. Fuente: Elaboración propia.
En la Tabla 1 y la figura 5 se puede apreciar que los mejores valores para la media los alcanzó el método Mean-Shift, con un valor promedio de 5.80. El resto de los métodos alcanzaron valores superiores: Level- Set obtuvo 6.57, Umbralización alcanzó 7.16 y Chan-Vese 7.36. Estos resultados son menos representativos debido a que se obtienen bordes menos cercanos a los aportados por los especialistas, y esto afecta la distancia mínima obtenida entre los pares de píxeles valorados. También en esta misma tabla y la figura 6 observamos que el método Mean-Shift obtuvo los mejores resultados, con una varianza promedio de 123.15. El resto de los métodos alcanzaron valores alejados a los mostrados por este método, Level-Set obtuvo un 174.89, Umbralización 182.72 y Chan-Vese 197.99, denotando una alta dispersión de los pixeles en comparación con el contorno obtenido por el método Mean-Shift.
En los resultados expuestos en la Tabla 2 y la figura 7 se observa que el método Mean Shift alcanzó los mejores resultados con una distancia de Baddeley de 8.24, seguido del método de Level-Set con 9.29, Umbralización 10.22 y Chan-Vese 10.57.
Teniendo en cuenta los resultados mostrados en las tablas 3 y las figuras 8 y 9 se observa que el método Mean-Shift alcanzó los segundos mejores resultados con valores de similitud de 0.74 para el índice de Jaccard y 0.85 para el índice de Dice, solo superado por el método de Umbralización con 0.78 y 0.88 respectivamente, en cuanto a la similitud de las regiones obtenidas con respecto a su respectiva región de prueba.
Teniendo en cuenta los resultados podemos determinar que Mean-Shift ofrece mejores respuestas debido a que alcanza el primer lugar para el primer grupo de métricas en las que es evaluada la distancia y la dispersión de los pixeles en los bordes de las imágenes obtenidas y además también obtiene buenos resultados en cuanto a la similitud de las regiones obtenidas solo superado en un pequeño margen por el algoritmo de Umbralización que alcanza un tercer lugar al analizar las primeras métricas.
Conclusiones
El presente trabajo propone un estudio experimental para evaluar la calidad de la segmentación en imágenes médicas de muestras de sangre periférica de pacientes con anemia drepanocítica obtenidas en los laboratorios clínicos del Hospital General Docente “Dr. Agostinho Neto” de la provincia de Guantánamo, utilizando métodos de segmentación Umbralización, Mean-Shift, Level-Set y Chan-Vese y medidas para evaluar la calidad de esta segmentación de media y varianza (valoración estadística), distancia de Baddeley (valoración geométrica) Jaccard y Dice (similitud entre regiones). La experimentación realizada permitió determinar que el método que mejor respuesta ofrece es el Mean-Shift, que alcanzó valores de media de 5.80, varianza de 123.15, distancia de Baddeley de 8.24, valores de similitud de 0.74 para el índice de Jaccard y 0.85 para el índice de Dice, superando en su conjunto al resto de los métodos de segmentación evaluados.